

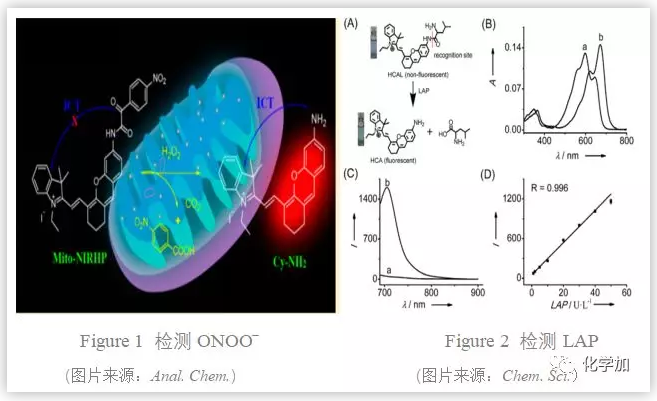
药物诱导的肝损伤(DILI)是典型的病理例子,该病理产生的主要原因就是药物引起的肝毒性。当发生DILI时亮氨酸氨肽酶(LAP)和活性氧/氮(ROS/RNS)的水平显著提高,因此LAP和ONOO−(一种ROS/RNS)水平可以作为早期诊断肝毒性的指标。基于以上原理,研究者开发出LAP和ONOO−响应的近红外探针来评价肝毒性。
传统的近红外探针存在化学结构不稳定的缺点,会产生假信号,导致不能准确评价肝毒性。当荧光探针稳定性较差时,探针在生物介质中识别出被分析物之前或之后,容易被生物相关分子(亲核或氧化物质)裂解,产生假阴性信号。到目前为止,大多数的努力都集中在提高探针本身的选择性来解决假信号。然而,在提高探针对体内反应性强的物质和亲核细胞的化学稳定性方面所作的努力较少。因此,合理设计高性能、高稳定的近红外荧光探针,实现对目标分析物的准确检测,避免对目标分析物产生虚假信号,对于实际应用至关重要。
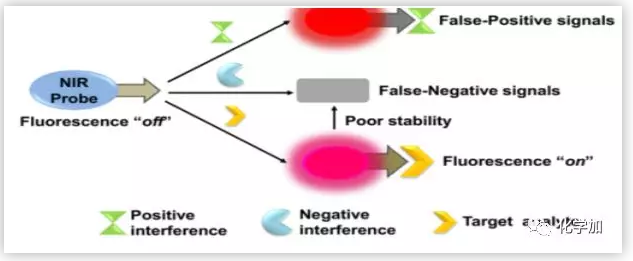
Figure 3 Probe响应示意(图片来源:J. Am. Chem. Soc.)
为了解决近红外探针在生理环境下产生假信号的问题,袁林团队对DCM(吡喃腈)、BODIPY、氰基、方酸、苯并吡喃等一系列近红外荧光染料在各种高活性物质(NO,ONOO−,O2•−,H2O2 ,H2S2 ,SO32−,SO3 2−)中的稳定性进行了测试,筛选出两种稳定的近红外荧光团(HD-NH2和compound 10)。在这两种荧光团的基础上,分别构建出用于监测LAP和ONOO的高保真荧光探针(NIR-LAP,NIR-ONOO−)。这两种探针均具有较高的灵敏度(LAP检测极限为80 mU/L, ONOO检测极限为90 nM),且对共存的ROS和RNS具有显著的稳定性。此外,通过细胞线粒体LAP和ONOO−的检测证实了探针的有效性,并用于观察LAP和ONOO−在药物诱导的肝毒性中的微小波动。最后,探针成功地用于监测扑热息痛(APAP,一种替代阿司匹林的解热镇痛药)诱导的肝毒性的补救药物的有效性。袁林团队的工作证明筛选与合理设计相结合的方法是改进高保真近红外探针的稳定性和灵敏度的一种方便有效的策略。
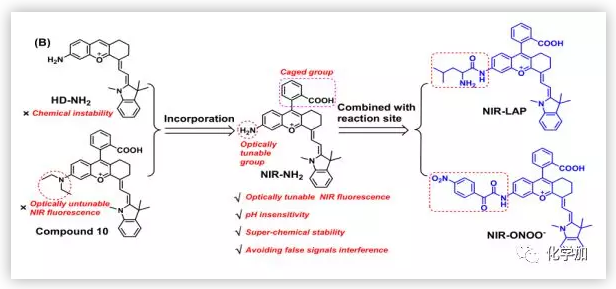
Figure 4设计思路(图片来源:J. Am. Chem. Soc.)
袁林团队对NIR-LAP,NIR-ONOO−的光谱分析发现:NIR-LAP在608/657 nm有强的吸收峰,但是荧光很弱,与LAP (0-100 U/L)反应后吸收峰红移到622/670 nm,703 nm的荧光增强;NIR-ONOO−与ONOO− (0-15 µM)反应后吸收从611/661 nm红移到627/678 nm并且荧光增强。这些结果表明NIR-LAP,NIR-ONOO−分别对LAP,ONOO−有高选择性(详情参见Supporting Information)。袁立团队接着测试了近红外探针NIR-LAP ,NIR-ONOO−在各种生物活性物质中对LAP,ONOO−的特异性和选择性,初步证明NIR-LAP ,NIR-ONOO−可以应用于生理条件下LAP,ONOO−的成像。
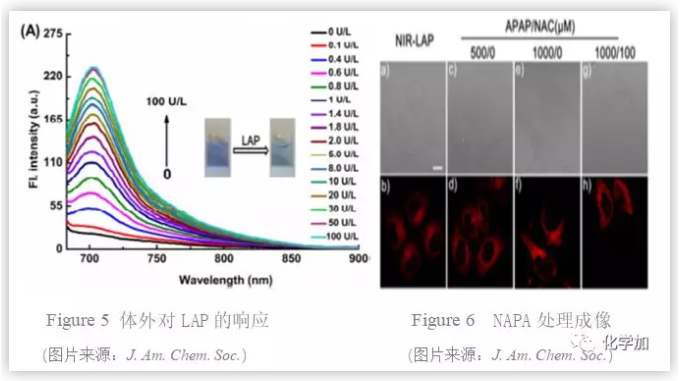
基于体外研究情况,作者进一步研究了在细胞内NIR-LAP,NIR-ONOO−检测LAP,ONOO−的情况,结果表明这两种近红外有很好的生物相容性和选择性,可以进行细胞成像。在此基础上,作者通过DILI模型细胞对两种探针进行了概念验证实验。在APAP诱导的肝损伤中,NIR-LAP,NIR-ONOO这两个近红外探针能够准确检测细胞内LAP和ONOO−的波动,并一步利用他们来研究活细胞和小鼠中肝毒性治疗与LAP或ONOO-水平之间的关系。作者以NAC为对照药物,选择谷胱甘肽(GSH)和葡萄糖内酯(Glu)作为肝保护药物在活HepG2细胞中进行实验。当LAP作为肝毒治疗指标时,通过比较NAC, GSH 和Glu诱导产生的荧光强度变化,可以得出这样的结论:NIR-LAP,NIR-ONOO−在评价出现DILI后进行的药物治疗是有效的,并可用于药物筛选和肝毒性成像的体内修复。
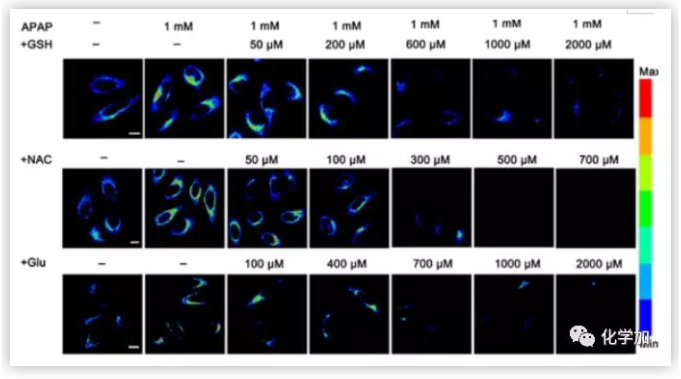
Figure 7 不同药物处理细胞的成像(图片来源:J. Am. Chem. Soc.)
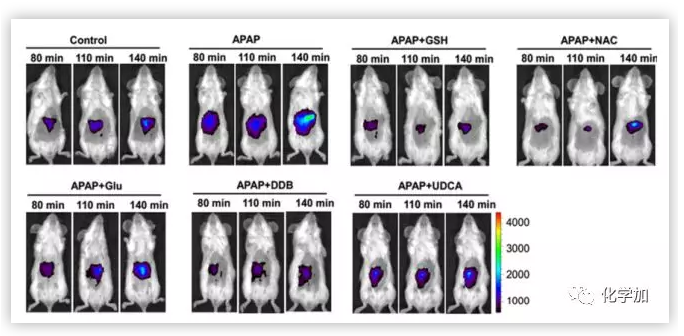
Figure 8 不同药物处理活体成像(图片来源:J. Am. Chem. Soc.)
总结:袁林团队通过对现有的近红外探针的筛选、整合,设计出光学可调高保真近红外探针。该探针可以准确、灵敏、选择性检测DILI,实现高保真活体成像,准确评价肝毒性。此外改变探针的识别位点,可以进行其他物质的分析和成像。这为未来药物评价和治疗中NIR荧光探针的设计提供了一种新的方法,同时也启发研究者如何在已有的研究结果上做出新意。
撰稿人:犟子柳
声明:化学加刊发或者转载此文只是出于传递、分享更多信息之目的,并不意味认同其观点或证实其描述。若有来源标注错误或侵犯了您的合法权益,请作者持权属证明与本网联系,我们将及时更正、删除,谢谢。 电话:18676881059,邮箱:gongjian@huaxuejia.cn